Breve historia
El ADN fue aislado por primera vez en 1869 por el médico Suizo Friedrich Miescher, quien realizaba experimentos acerca de la composición química de la pus. Al examinar las vendas con pus, notó un precipitado de una sustancia desconocida la cual llamó nucleína, debido a que lo había extraído a partir de los núcleos.4 Sin embargo, su estructura y carácter químico aún eran completamente desconocidos.
En 1919 Phoebus Levene identificó que un nucleótido está formado por una base nitrogenada, un azúcar y un grupo fosfato. También identificó la existencia del ADN y del ARN. En 1928, Frederick Griffith trabajaba con cepas lisas y rugosas de una bacteria, las cuales tenían un “factor transformante”; fragmentó la nucleína, y de ella separó un componente proteico y un grupo prostético, este último, por ser un ácido, le llamó ácido nucleico.4
Años más tarde, en 1930 Levene y su maestro Albrecht Kossel determinaron que la nucleína de que había identificado Miescher era el ácido desoxirribonucleico ADN formado por cuatro bases nitrogenadas adenina, guanina, citosina y timina, el azúcar desoxirribosa y un grupo fosfato.4 En 1937 William Astbury produjo el primer patrón de difracción de rayos x que mostraba que el ADN tenía una estructura regular.4
En 1944, Oswald Avery, Colin MacLeod y Maclyn McCarty extrajeron la fracción activa del “factor transformante” y, mediante varios análisis, observaron que estaba constituido principalmente por “una forma viscosa de ácido desoxirribonucleico que no contenía proteínas, lípidos, ni polisacáridos.4
En 1952 Alfred Hershey y Martha Chase comprobaron que un fago transmitía su información genética en su ADN, pero no su proteínas. El bioquímico austriaco Erwin Chargaff analizó diferentes especies de ADN determinó que la adenina, tiamina, citosina y guanina no se encontraban en cantidades iguales, variaban entre especies, pero no de entre individuos de la misma especies.1
En 1953 Rosalind Franklin, química que estudiaba la estructura de las moléculas con la técnica de cristalografía de rayos X, se enfocó en utilizar esa técnica para estudiar el ADN.6 Maurice Wilkins, Francis Crick y James Watson lograron mediante estos estudios de difracción de rayos X, descubrir la estructura molecular de doble hélice del ADN, por lo que les otorgaron el premio Novel de fisiología y medicina en 1962.4 Rosalind Franklin falleció antes de su reconocimiento 1
Desde 1960 se estuvo intentando con renacuajos y otros animales la clonación artificial, hasta en 1997 Wilmut: permitió demostrar que las manifestaciones fenotípicas vienen del ADN con la oveja Dolly, que no fue clonación, sino fue creada a partir de una célula somática.1
Introducción
ADN y ARN
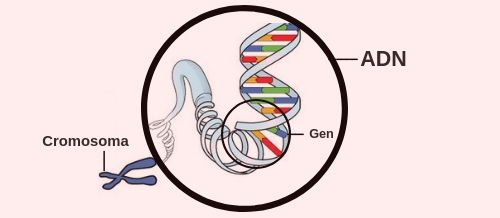
Los dos tipos principales de ácidos nucleicos son el ácido desoxirribonucleico (ADN) y el ácido ribonucleico (ARN).4 El ADN es el material genético que se encuentra en todos los organismos vivos, en el núcleo de las células eucariotas y en los orgánulos, cloroplastos y mitocondrias, también forma un complejo con las proteínas histonas para formar la cromatina, que forma el cromosoma y estos puede contener miles de genes (ver figura 1). Estos genes contienen la información para hacer proteínas para la mayoría de las funciones de nuestro cuerpo.4
Las moléculas de ADN nunca abandonan el núcleo, sino que utilizan un intermediario para comunicarse con el resto de la célula. El ARN, participa principalmente en la síntesis de proteínas. Este intermediario es el ARN mensajero (ARNm). Otros tipos de ARN, como rRNA, tRNA y micro RNA, están involucrados en la síntesis de proteínas y su regulación.4 El ADN y el ARN están formados por monómeros conocidos como nucleótidos (ver figura 1).
En general, una base ligada a un azúcar se denomina nucleosido y una base ligada a un azúcar y a uno o más grupos fosfatos recibe el nombre de nucleotido 14
Componentes
Acido fosfórico
Su fórmula química es H3PO4. Cada nucleótido puede contener; un monofosfato AMP. dos difosfato ADP, o tres trifosfato ATP. aunque como monómero constituyentes de los ácidos nucleicos solo aparecen en forma de nucleósidos monofosfatos. 14 (ver figura 2).

Azúcar Desoxirribosa y Ribosa
La estructura de soporte de una hebra de ADN está formada por unidades de grupos fosfato y azúcar desoxirribosa. 14
El azúcar en el ADN es la desoxirribosa, (ver figura 3). y en el ARN el azúcar es la ribosa. (ver figura 4). La diferencia entre los azúcares es la presencia del grupo hidroxilo en el segundo carbono. 4
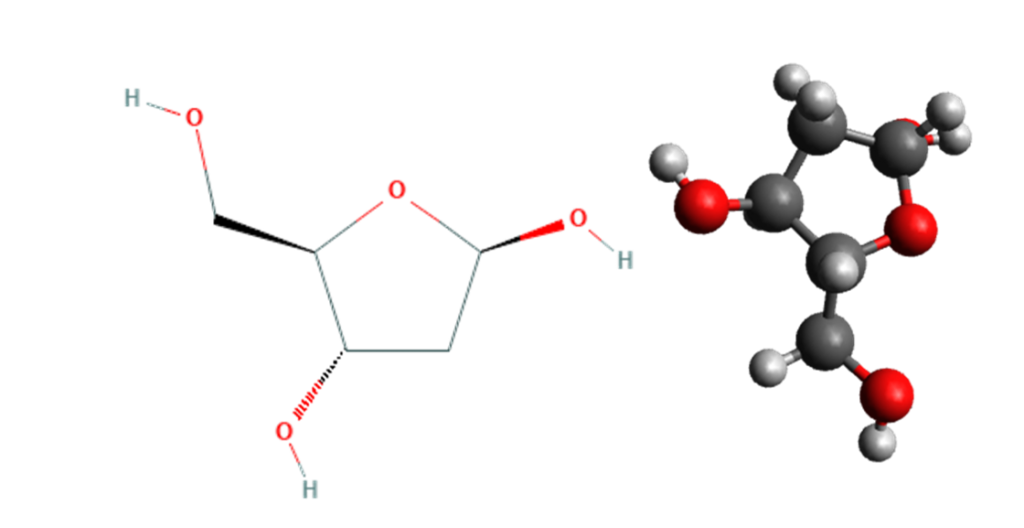
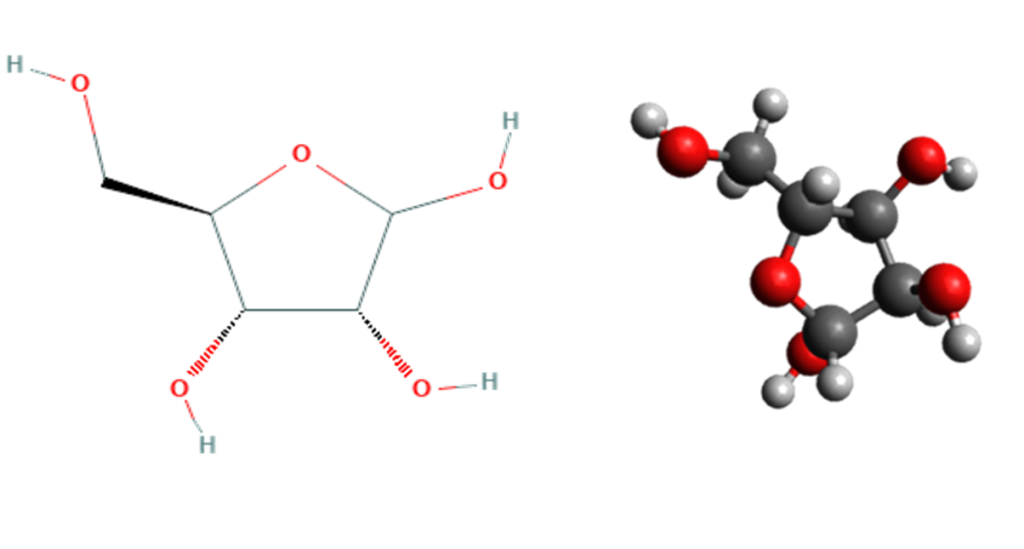
Los átomos de carbono de la molécula de azúcar están numerados como 1 ‘, 2’, 3 ‘, 4’ y 5 ‘(lee como “un primo”).7 Las moléculas de azúcar se unen entre sí a través de grupos fosfato, que forman enlaces fosfodiéster.(ver figura 5). Entre los átomos de carbono tercero (3′, «tres prima») y quinto (5′, «cinco prima») de dos anillos adyacentes de azúcar.14

En toda la cadena del ácido nucleico, los enlaces fosfodiester presentan la misma orientación, por lo que existe una polaridad de la hebra. En una doble hélice, la dirección de los nucleótidos en una hebra (3′ → 5′) es opuesta a la dirección en la otra hebra (5′ → 3′). Esta organización de las hebras de ADN se denomina anti paralela; son cadenas paralelas, pero con direcciones opuestas. La estructura de los ácidos nucleicos se representa con el extremo 5′ a la izquierda y el 3′ a la derecha.8
Bases Nitrogenadas
Las bases nitrogenadas, son moléculas orgánicas que contienen carbono y nitrógeno y son bases porque contienen un grupo amino que puede unir un hidrógeno adicional y disminuye la concentración de iones de hidrógeno a su alrededor, haciéndolo más básico. 4 Las bases tienen compuestos heterocíclicos y aromáticos con dos o más átomos de nitrógeno, se clasifican en dos grupos;
Bases púricas
Bases púricas o purinas adenina (ver figura 11) y guanina (ver figura 12), derivadas de la purina y formadas por dos anillos unidos entre sí, carbono-nitrógeno, su anillo formado no es totalmente plano.9
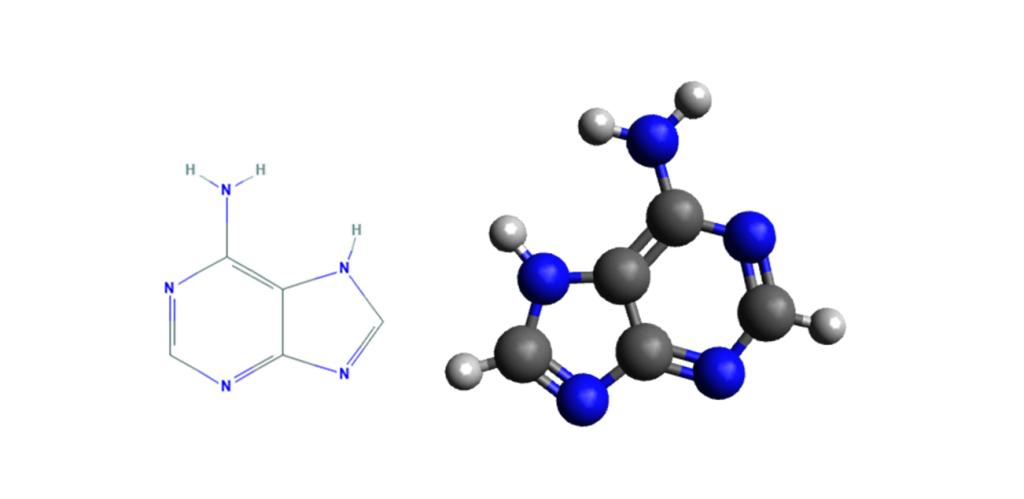
Adenina
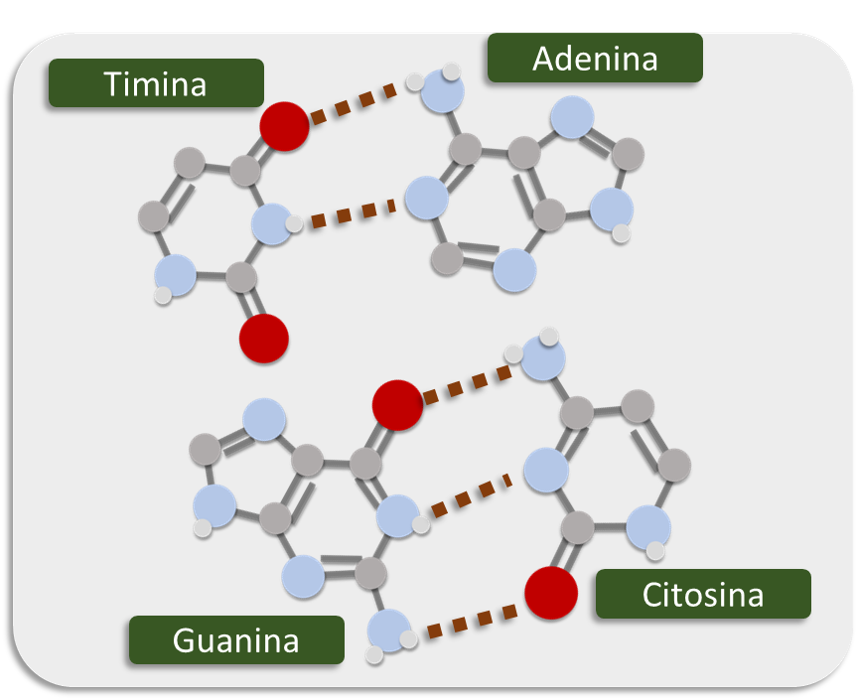
En el código genético se representa con la letra A. Es un derivado de la purina con un grupo amino en la posición 6. Forma el nucleósido adenosina(desoxiadenosina en el ADN) y el nucleótido adenilatoo (desoxi)adenosina monofosfato (dAMP, AMP). En el ADN siempre se empareja con la timina de la cadena complementaria mediante 2 puentes de hidrógeno, A=T. Su fórmula química es C5H5N5 y su nomenclatura 6-aminopurina. La adenina, junto con la timina, fue descubierta en 1885 por el médico alemán Albrecht Kossell También forma parte de la molécula de trifosfato de adenosina, que constituye la fuente principal de energía a nivel celular, y está presente en muchas sustancias naturales como la remolacha, el té y la orina.17, (ver figura 11). 18
Guanina
En el código genético se representa con la letra G. Es un derivado púrico con un grupo oxo en la posición 6 y un grupo amino en la posición 2. Forma el nucleósido (desoxi)guanosina y el nucleótido guanalato o (desoxi)guanosina monofosfato (dGMP, GMP). La guanina siempre se empareja en el ADN con la citosina de la cadena complementaria mediante tres enlaces de hidrógeno, G≡C. Su fórmula química es C5H5N5O y su nomenclatura 6-oxo, 2-aminopurina. (ver figura 12).18
Bases pirimidínicas
Bases pirimidínicas o bases pirimidinas citosina(ver figura 13). y timina(ver figura 14), derivadas de la pirimidina y con un solo anillo. El anillo de las pirimidinas son anillos aromáticos heterocíclicos con seis miembros y con dos átomos de nitrógeno, es un sistema plano, Los átomos son numerados siguiendo el sentido de las agujas del reloj.9
Citosina
En el código genético se representa con la letra C. Es un derivado pirimidínico, con un grupo amino en posición 4 y un grupo oxo en posición 2. Forma el nucleosido citidina(desoxicitidina en el ADN) y el nucleotido citililato o (desoxi)citidina monofosfato (dCMP en el ADN, CMP en el ARN). La citosina siempre se empareja en el ADN con la guanina de la cadena complementaria mediante un triple enlace, C≡G. Su fórmula química es C4H5N3O y su nomenclatura 2-oxo, 4 aminopirimidina. 18 Su masa moleculares de 111,10 unidades de masa atómica. La citosina se descubrió en 1894, al aislarla del tejido del timo de carnero.16 (ver figura 13).9
En los ácidos nucleicos existe una quinta base pirimidínica, denominada uracilo (U), que normalmente ocupa el lugar de la timina en el ARN y difiere de esta en que carece de un grupo metilo en su anillo. El uracilo no se encuentra habitualmente en el ADN, solo aparece raramente como un producto residual de la degradación de la citosina por procesos de desaminación oxidativa 15 Entre el anillo de imidazol y el anillo de las pirimidinas se ha reportado un ligero pliegue o arruga.9
Timina
En el código genético representa con la letra T. Es un derivado pirimidínico con un grupo oxo en las posiciones 2 y 4, y un grupo metil en la posición 5. Forma el nucleosido timina y el nucleotido timidilato o timidina monofosfato (dTMP). 18 En el ADN, la timina siempre se empareja con la adenina de la cadena complementaria mediante 2 puentes de hidrógeno, T=A. Su fórmula química es C5H6N2O2 y su nomenclatura 2, 4-dioxo, 5-metilpirimidina. 9 ,15

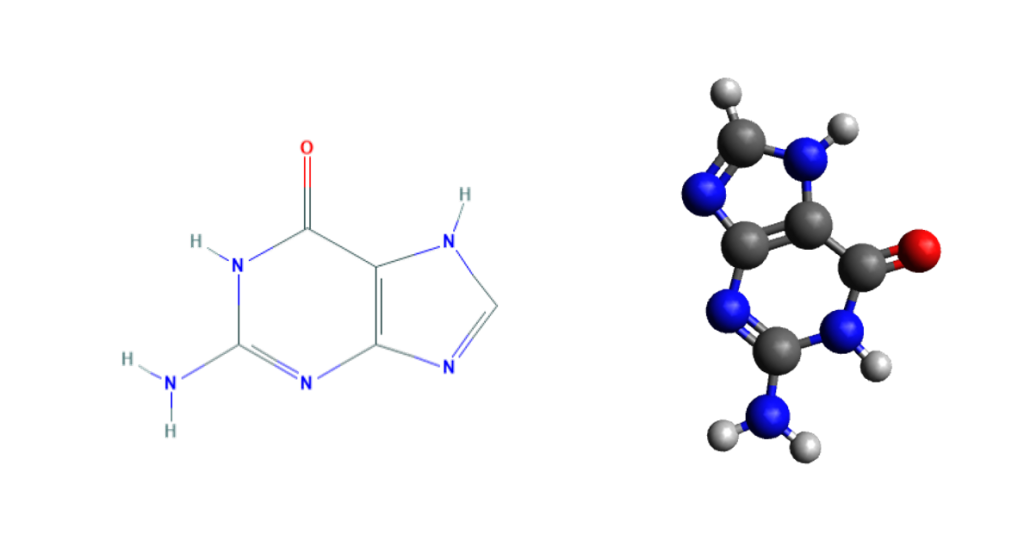
También existen otras bases nitrogenadas llamadas bases nitrogenadas minoritarias, derivadas de forma natural o sintética de alguna otra base mayoritaria. Lo son por ejemplo la hipoxantina , relativamente abundante en el tARN , o la cafeina (ver figura 15). Ambas derivadas de la adenina; otras, como el aciclovir, derivadas de la guanina, son análogos sintéticos usados en terapia antiviral; otras, como una de las derivadas del uracilo, son antitumorales.18
Propiedades de las bases Nitrogenadas
Aromaticidad
En química orgánica, un anillo aromático se define como una molécula cuyos electrones de los enlaces dobles tienen libre circulación dentro de la estructura cíclica. La movilidad de los electrones dentro del anillo le da estabilidad a la molécula.9.
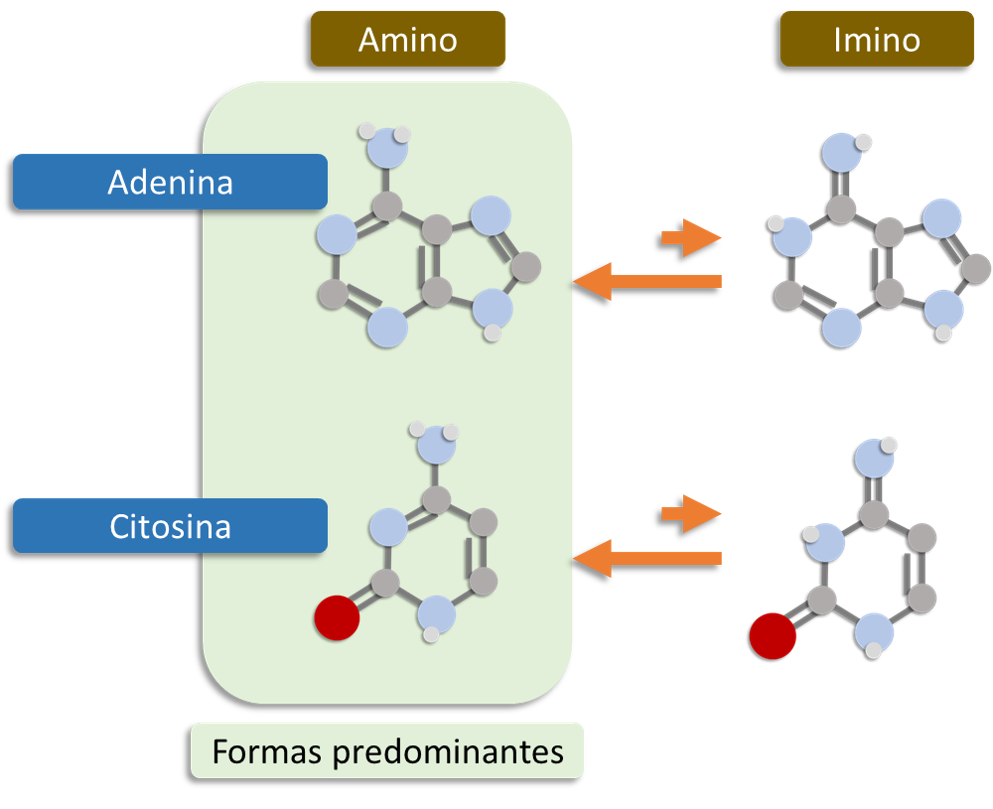
Tautomeria o isomería
Debido a que un átomo de hidrógeno unido a otro átomo puede migrar a una posición vecina en las bases nitrogenadas se dan dos tipos de tautomería. 18
Tautomería lactama-lactima, donde el hidrógeno migra del nitógeno al oxigeno del grupo oxo forma lactama y visceversa forma lactima.18. Tautomeria imina-amina, donde el hidrógeno puede estar formando el grupo amina o migrar al nitógeno adyacente forma imina.18
La adenina sólo puede presentar tautomería amina-imina, la timina y el uracilo muestran tautomería doble lactama-lactima, y la guanina y citosina pueden presentar ambas (ver figura 16). Por otro lado, y aunque se trate de moléculas apolares, las bases nitrogenadas presentan suficiente carácter polar como para establecer puentes de hidrógeno ya que tienen átomos muy electronegativos (nitrógeno y oxígeno) que presentan carga parcial negativa, y átomos de hidrógeno con carga parcial positiva, de manera que se forman dipolos que permiten que se formen estos enlaces débiles .18
La naturaleza aromática de este sistema de anillos les otorga la capacidad de experimentar un fenómeno llamado tautomería ceto-enol.9 Es decir, las purinas y las pirimidinas existen en pares tautoméricos. Los tautómeros ceto son predominantes a pH neutro para las bases uracilo, timina y guanina. En contraste, la forma enol es predominante para la citosina, en pH neutros. Este aspecto es fundamental para la formación de puentes de hidrógeno entre las bases.9
Absorción de luz UV
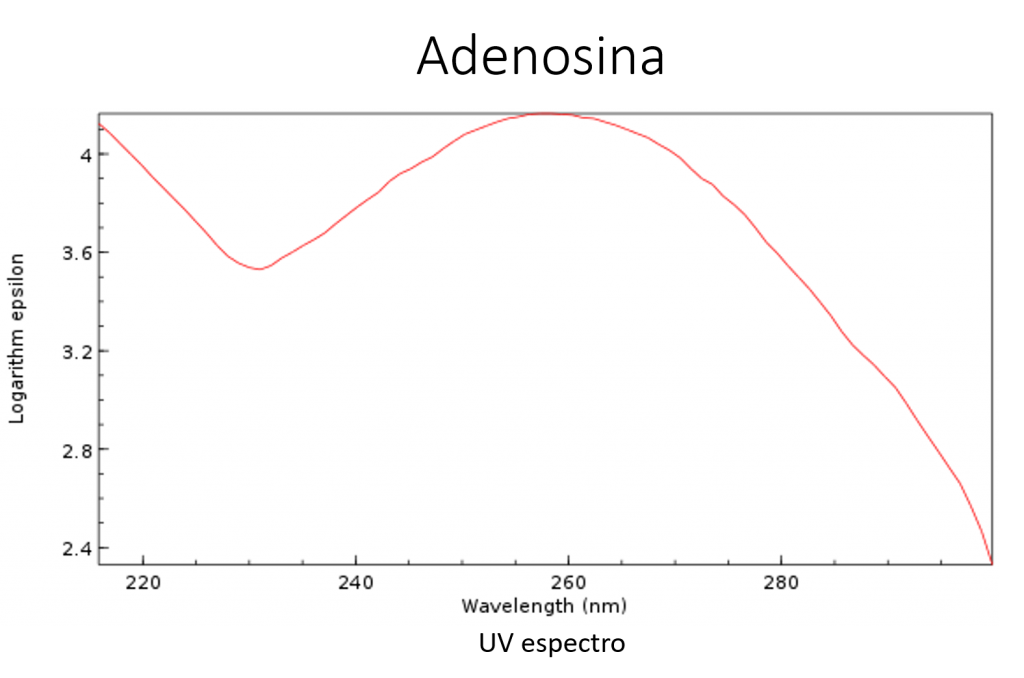
Otra propiedad de las purinas y las pirimidinas es su capacidad de absorber fuertemente la luz ultravioleta (luz UV). Este patrón de absorción es una consecuencia directa de la aromaticidad de sus anillos heterocíclicos.El espectro de absorción tiene un máximo cercano a los 260 nm. (ver figura 17).Propiedad que sirve para cuantificar la cantidad de ADN en muestras.9
Solubilidad en agua.
Gracias al fuerte carácter aromático de las bases nitrogenadas, estas moléculas son prácticamente insolubles en el agua.9
Apareamiento de bases
La dóble hélice de ADN se mantiene estable mediante la formación de puentes de hidrógeno. Para la formación de un enlace de hidrógeno una de las bases debe presentar un “donador” de hidrógenos con un átomo de hidrógeno con carga parcial positiva (-NH2 o -NH) y la otra base debe presentar un grupo “aceptor” de hidrógenos con un átomo cargado electronegativamente( C=O o N). 18.Los puentes de hidrógeno no son enlaces covalentes, pueden romperse y formarse de nuevo de forma relativamente sencilla. Por esta razón, las dos hebras de la doble hélice pueden separar por fuerza mecánica o por alta temperatura.19. Los dos tipos de pares de bases forman un número diferente de enlaces de hidrógeno: A=T. forman dos puentes de hidrógeno, y C≡G figura tres puentes de hidrógeno . El par de bases GC es por tanto más fuerte que el par de bases AT.(ver figura 18). Las dobles hélices largas de ADN con alto contenido en GC tienen hebras que interaccionan más fuertemente que las dobles hélices cortas con alto contenido en AT.19
Estructura del ADN
Estructura primaria
La diferencia de la información radica en la distinta secuencia de bases nitrogenadas. Esta secuencia presenta un código, que determina según el orden de las bases.18
Estructura secundaria
En una cadena doble, destrógira o levógira, son complementarias, son antiparaleas pues el extremo 3´ se enfrenta al extremo 5′. Existen tres modelos de ADN, el de tipo B es el más abundante y es el que describe la estructura descrita por Watson y Crick .18
Estructura terciaria
Es como se almacena el ADN en un espacio reducido, para formar los cromosomas. Varía según se trate de organismos procariotas o eucariotas. En células procariotas el ADN se pliega como una super-hélice, en forma circular, asociada a una pequeña cantidad de proteínas, organelos celulares como mitocondrias y en los cloroplastos. 18 En células eucariotas la cantidad de ADN en cada cromosoma es muy grande se requieren proteínas como las histonas y proteínas no histonicas como las protaminas.17
Estructura cuaternaria
El enrollamiento de los nucleosomas recibe el nombre de solenoide. Dichos solenoides se enrollan formando la cromatina del núcleo interfásico de la célula eucariota. Cuando la célula entra en división, el ADN se compacta más, formando así los cromosoma.18
Hendiduras mayor y menor
La doble hélice es una espiral dextrógira, esto es, cada una de las cadenas denucleotidos gira a derechas; yendo de abajo a arriba, si las dos hebras giran a derechas se dice que la doble hélice es dextrógira, y si giran a izquierdas, levógira la conformación más común que adopta el ADN, la doble hélice es dextrógira, girando cada par de bases respecto al anterior unos 36º.
Cuando las dos hebras de ADN se enrollan una sobre la otra, se forman huecos o hendiduras entre una hebra y la otra, dejando expuestos los laterales de las bases nitrogenadas del interior. En la conformación más común que adopta el ADN aparecen, como consecuencia ángulos formados entre los azucares de ambas cadenas de cada par de bases nitrogenadas formadose , dos hendiduras alrededor, la hendidura o surco mayor, que mide 22 Å A(2,2 nm) de ancho, y la otra, la hendidura o surco menor, que mide 12 Å (1,2 nm) de ancho. Cada vuelta de hélice, cuando esta ha realizado un giro de 360º de principio de hendidura mayor a final de hendidura menor, medirá por tanto 34 Å. (ver figura 19).

La anchura de la hendidura mayor implica que los extremos de las bases son más accesibles en esta, de forma que la cantidad de grupos químicos expuestos también es mayor lo cual facilita la diferenciación entre los pares de bases A-T, T-A, C-G, G-C. también se verá facilitado el reconocimiento de secuencias de ADN por parte de diferentes proteínas sin la necesidad de abrir la doble hélice.
Las proteínas como los factores de transcripción que pueden unirse a secuencias específicas, frecuentemente contactan con los laterales de las bases expuestos en la hendidura mayor, los grupos que quedan expuestos en la hendidura menor son similares, de forma que el reconocimiento de los pares de bases es más difícil; por ello se dice que la hendidura mayor contiene más información que la hendidura menor.24
Estructuras en doble hélice.
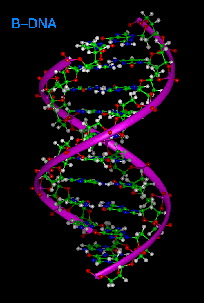
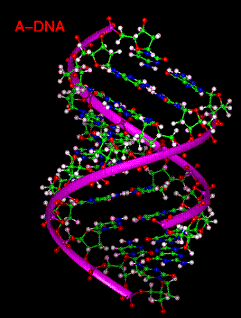
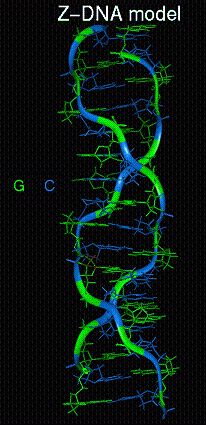
El ADN existe en muchas conformaciones ,en organismos vivos sólo se han observado las conformaciones ADN-A,ADN-B y ADN-Z La conformación que adopta el ADN depende de su secuencia, la cantidad y dirección de superenrollamiento que presenta, la presencia demodificaciones químicas en las bases y las condiciones de la solución, tales como la concentración de iones de metales y poliaminas .20 De las tres conformaciones, la forma “B” es la más común en las condiciones existentes en las células. Las dos dobles hélices alternativas del ADN difieren en su geometría y dimensiones.
ADN-B
Las figuras siguientes son representaciones de la estructura B-ADN. 13. la conformación del anillo de azúcar: C3′-endo, el número de nucleótidos por vuelta son 10 , la conformación del enlace glucosídico en su ángulo de torsión es de 36º13, En esta estructura la profundidad del surco mayor es muy similar a la del surco menor, debido a que las bases que se aparean son prácticamente coplanares y a que el eje de la hélice corta a este plano de una forma casi perpendicular, pasando además por el centro del par de bases. 13. (ver figura 20).
ADN-A o RNA-11
La forma “A” ocurre en condiciones no fisiológicas en formas deshidratadas, cuando se reduce la humedad relativa por debajo de 75%, las vueltas son más cortas, hay más nucleótidos por vuelta, las bases están más inclinadas , con una hendidura menor superficial y más amplia, y una hendidura mayor más estrecha y profunda. No hay ningún hueco posible para el agua, los fosfatos están más cerca unos de otros en vertical. mientras que en la célula puede producirse en apareamientos híbridos de hebras ADN-ARN, esta estructura no se ha encontrado in vivo pero la adoptan los híbridos ADN-ARN y el ARN bicatenario. 12
En el A-ADN el eje de la hélice no pasa por el centro del par de bases, sino que forma con el plano de estas un ángulo de entre 13 y 19º, lo que hace que el surco mayor sea muy profundo y que se extienda desde la superficie hacia el interior sobrepasando el eje central y adentrándose en la otra mitad del cilindro helicoidal, por el contrario, el surco menor es muy poco profundo, sólo una pequeña depresión en el cilindro helicoidal.13( ver figura 21).
ADN- Z
Los segmentos de ADN en los que las bases han sido modificadas por metilacion pueden tener cambios conformacionales mayores y adoptar la forma “Z”. En este caso, las hebras giran alrededor del eje de la hélice en una espiral que gira a mano izquierda, lo opuesto a la forma “B” más frecuente.Esta estructura se favorece por la alternancia de purinas y pirimidinas se ha comprobado que el ADN-Z existe in vivo por anticuerpos desarrollados contra el ADN-Z12, su estructura es más estrecha, más larga, hay más nucleótidos por vuelta, surco mayor superficial y surco menor profundo. Estas estructuras poco frecuentes pueden ser reconocidas por proteínas específicas que se unen a ADN-Z y posiblemente estén implicadas en la regulación de la transcripción 21 El ADN.Z es una forma estructural observada in vitro en segmentos en donde se da la secuencia alternante dG-dC y en altas concentraciones salinas o cuando se añade alcohol. (ver figura 21).
ADN cuádruplex
En los extremos de los cromosomas lineales existen regiones especializadas de ADN denominadas telómeros . La función principal de estas regiones es permitir a la célula replicar los extremos cromosómicos utilizando la enzima telomerasa , porque las enzimas que replican el resto del ADN no pueden copiar los extremos 3′ de los cromosomas. Estas terminaciones cromosómicas especializadas también protegen los extremos del ADN, y evitan que los sistemas de reparación del ADN en la célula los procesen como ADN dañado que debe ser corregido. ( ver figura 22). En las células humanas, los telómeros son largas zonas de ADN de hebra sencilla que contienen algunos miles de repeticiones de una única secuencia TTAGGG.11,22
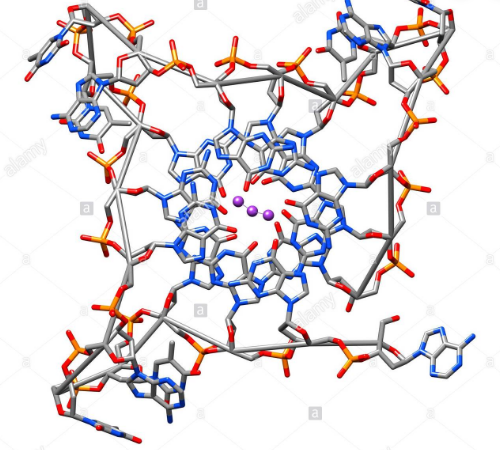
Estas secuencias con guanina pueden estabilizar los extremos cromosómicos con la formación de estructuras de cuatro bases las cuatro bases de guanina forman unidades con superficie plana que se apilan una sobre otra, y forman una estructura cuádruple-G 22.
También se pueden formar otras estructuras, con el juego central de cuatro bases, con una una hebra sencilla plegada alrededor de las bases, o varias hebras paralelas diferentes, de forma que cada una contribuye con una base a la estructura central.
Los telómeros también forman largas estructuras en lazo, denominadas lazos teloméricos o lazos-T (T-loops en inglés), las hebras simples de ADN se enroscan sobre sí mismas en un círculo estabilizado por proteínas que se unen a telómeros. En el extremo del lazo T, el ADN telomérico de hebra sencilla se sujeta a una región de ADN de doble hebra porque la hebra de ADN telomérico altera la doble hélice y se aparea a una de las dos hebras. Esta estructura de triple hebra se denomina lazo de desplazamiento o lazo D (D-loop).23
i-motif
El i-motif son estructuras secundarias de ADN de cuatro cadenas que se forman en un punto particular en el ciclo de vida de las células, en la última fase del G1, cuando el ADN es leído activamente,10,(ver figura 23), se cree que se forman en regiones del genoma ricas en citosina y tienen funciones reguladoras Son estabilizados por condiciones ácidas, se componen de dos cadenas duplex de ADN de cadena paralela mantenidos juntos en una orientación anti-paralela por pares de bases citocina -citosina intercalados y dependen del ciclo celular, de factores como la secuencia y las condiciones ambientales 14
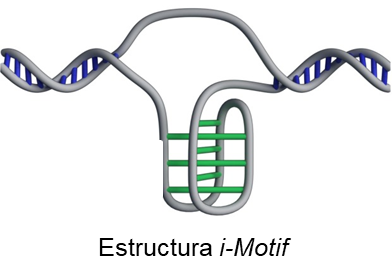
También los i-motifs aparecen en algunas regiones relacionadas con la regulación genética en las que el ADN controla si los genes son activados o no y se pongan en marcha determinados mecanismos moleculares. El fallo de estos mecanismos puede tener consecuencias desfavorables y en los telómeros, los extremos de los cromosomas son importantes para el proceso de envejecimiento. 10
El ADN no codificante
El ADN no codificante corresponde a secuencias del genoma que no generan una proteína, pero regulan genes para determinar cuántos genes se tienen que activar; como se debe empacar el ADN en los cromosomas, 35 por ejemplo los telómero y centrómeros contienen pocos o ningún gen codificante de proteínas, pero son importantes para estabilizar la estructura de los cromosomas. 25
Enzimas que modifican el ADN
Nucleasas
Las nucleasas son enzimas que cortan las hebras de ADN mediante la hidrólisis de enlaces fosfodiéster. Las exonucleasas fragmentan las hebras de ADN en los extremos de la secuencia mientras que las endonucleasas cortan el ADN por secuencias específicas en el interior de las hebras y se utilizan con mayor frecuencia y las que se utilizan son las enzimas de restricción. 29
Ligasas.
Las ADN ligasas pueden reunir hebras de ADN separadas realizando la reparación del ADN. 29
Topoisomerasas
Las topoisomeras son enzimas que tienen a la actividad de nucleasas y ligasas. Estas enzimas cortan la hélice de ADN reduciendo el grado de superenrollamiento y vuelven a unir el ADN. Otras enzimas cortan una hélice de ADN y luego pasan a una segunda hebra de ADN a través de la rotura, antes de reunir las hélices, están presentes en los procesos de replicación del ADN y de la transcripción.29
Helicasas.
Las helicasas utilizan energía almacenada en los nucleósidos trifosfatos ATP, para romper puentes de hidrógeno entre bases y separar la doble hélice de ADN en hebras simples. Estas enzimas son importantes para la mayoría de los procesos en los que las enzimas necesitan acceder a las bases del ADN.
Polimerasas
Las polimerasa son enzimas que sintetizan cadenas de nucleótidos a partir de nucleósidos trifosfatos. Su secuencia son copias de cadenas de polinucleótidos que ya existen y se denominan moldes, su función es ir añadiendo nucleótidos al grupo hidroxilo en la posición 3′. Todas las polimerasas funcionan en dirección 5′ –> 3′.30 Existen varias polimerasas:
Polimerasa dependiente de ADN
Una polimerasa realiza una copia de ADN a partir de otra secuencia de ADN, pueden reconocer errores en la síntesis y generan un desacoplamiento, por lo activan a una exonucleasa. 30
Polimerasa dependiente de ARN.
Las polimerasas dependientes de ARN hacen copias de la secuencia de una hebra de ARN en ADN, ejemplos; la telomerasa es una polimerasa que contiene su propio molde de ARN como parte de su estructura,31 y la transcriptasa reversa se une a una secuencia del ADN denominada promotor, y separa las hebras del ADN, copia la secuencia del gen en un transcrito de ARN mensajero hasta alcanzar una región de ADN denominada terminador donde se detiene y se separa del ADN.
Daño del ADN
El ADN pude ser dañado por mutágeno por ejemplo; la luz ultravioleta daña al ADN produciendo dímero de timina, radicales libre y el peróxido de hidrógeno producen modificaciones de bases en la guanina y roturas de dobles hebras, estas últimas, son las más difíciles de reparar y pueden producir mutaciones puntuales, inserciones y deleciones de la secuencia de ADN así como translocaciones cromosómicas 26 Muchos mutágenos se encuentran entre dos pares de bases, del ADN, las separa y cambia la doble hélice , pueden inhiben la transcripción y replicación del ADN, estos mutágenos se utilizan para inhibir el crecimiento de las células cancerosas, se les denomina agentes intercalantes, y pueden ser moléculas aromáticas,27 como;
Bromuro de etidio, su fórmula C21H20BrN3. Es un compuesto fluorescente, aromático, con una estructura tricíclica con grupos amino-benceno en cada lado tiene una molécula piridínica con seis átomos de nitrógeno y un anillo aromático. (ver figura 24).

Clorhidrato de daunomicina es un fármaco que bloquea la multiplicación celular y la reparación del ADN por la inhibición de la topoisomerasa, su nombre comercial es el cerubidine.
La doxorubicina, es un fármaco de la familia de las antraciclinas, y es una molécula intercalante del ADN, su nombre comercial Adriamicina o Rubex, el nuevo fármaco es Doxil que tiene liposomas encapsulados.
Benzopireno pertenece a una clase de hidrocarburos aromáticos tiene 2 o más anillos bencénicos, ya sea en forma simple o múltiple, formando cadenas o racimos.
Acridinas, C13H9N, es un compuesto orgánico y un compuesto heterocíclico del nitrógeno. La acridina se obtiene a partir de los aceites de antraceno, y por el proceso de destilación del alquitrán, se utiliza en la fabricación de colorantes.
Aflatoxinas son toxinas producidas por ciertos hongos en el maíz, el maní o cacahuates, la semilla de algodón y los frutos secos. son producidas principalmente por Aspergillus flavus y Aspergillus parasiticus, las aflatoxinas son carcinógenos potentes que pueden afectar a cualquier órgano o sistema, y especialmente al hígado y el riñón.
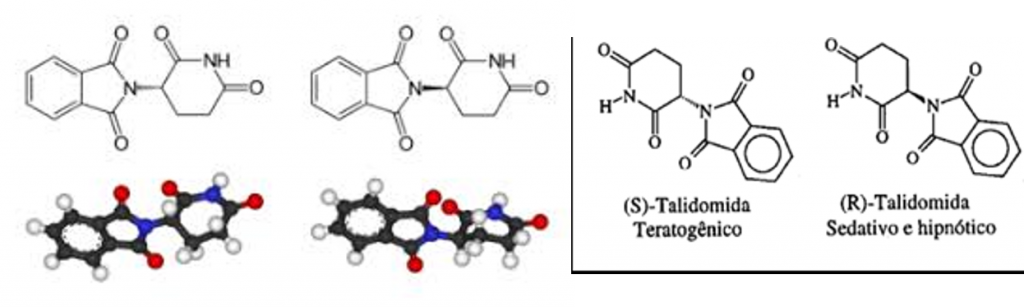
Talidomida es un compuesto con C13H10N2O4 tiene dos enantiómeros la forma R produce un efecto sedante y la forma S produce un efecto teratogénico. (ver figura 25), En los años 60 se utilizó como un fármaco sedante y para náuseas en mujeres embarazadas y posteriormente se descubrió que producía mutaciones causando malformaciones congénitas, En los últimos años se ha descubierto propiedades antiinflamatorias, e inmunoreguladoras.
El daño en el ADN puede provocar la alteración de procesos fisiológicos, como síntesis, transporte y degradación de proteínas. si el daño es grande y no puede ser reparado, lleva consigo la muerte celular.26
Estructura del ARN
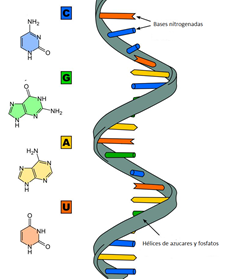
El ácido ribonucleico, o ARN, participa principalmente en el proceso de síntesis de proteínas bajo la dirección del ADN. El ARN generalmente es de cadena sencilla y está hecho de ribonucleótidos que están unidos por enlaces fosfodiéster, (ver figura 26). Un ribonucleico en la cadena de ARN contiene ribosa (el azúcar pentosa), una de las cuatro bases nitrogenadas (A, U, G y C) y el grupo fosfato. Existen cuatro tipos principales de ARN: ARN mensajero, , ARN de transferencia, ARN ribosómico y microARN (miARN).
El ARN mensajero.
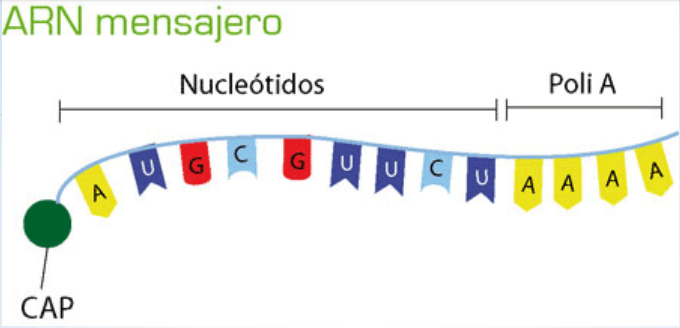
El ARN mensajero presenta una estructura lineal de una sola hebra. Su función es trasladar la información genética del ADN a los ribosomas, para la síntesis de proteínas. Cada molécula de ARNm es complementaria a un fragmento o gen de ADN, que sirve de molde para su síntesis durante la transcripción7. La poli A es una cola de adeninas de 80 a 250 residuos de adenina se le llama una Poliadenilación, se adhiere al segmento 3’ y ayuda a que el ARNm no se degrade en el citoplasma, por ribonucleasa, Por el otro lado en el segmento de 5’ se añade durante la transcripción un gorro o CAP que es una guanina modificada que es un residuo de 7–metilguanosina anclado al extremo por un enlace del tipo 5′,5’–trifosfato, (ver figura 27). El CAP al igual que la Poli A aumenta la vida del ARNm en el citoplasma a demás de que los ribosomas reconocerán a ese ARNm para mediar la traducción a proteínas.
Esta estructura se conoce como “cola” poliA y sirve como zona de unión para varias proteínas. Cuando un procariota adquiere una cola poliA tiende a estimular su degradación
ARNm monocistrónico: contiene la información necesaria para la síntesis de una proteína en eucariotas.7 ARNm policistrónico: contiene información para la síntesis de varias proteínas procariotas (ver figura 28).7

Splicing de ARN
El splicing de ARN o proceso de corte y empalme que consiste en la eliminación de los intrones en el ARN mensajero. Los intrones son secuencias de ADN que no son parte del gen pero que “interrumpen” dicha secuencia. Los intrones no se traducen y por ello deben ser eliminados del mensajero, algunos intrones encontrados en genes nucleares o mitocondriales pueden realizar el proceso de splicing sin ayuda de enzimas o ATP y se lleva a cabo por reacciones de transesterificación. El proceso de splicing es mediado por un complejo proteico denominado espliceosoma o complejo de corte y empalme (ver figura 29). El sistema está integrado por complejos de ARN especializados llamados ribonucleoproteínas pequeñas nucleares (RNP). Existen cinco tipos de RNP: U1, U2, U4, U5 y U6, que se encuentran en el núcleo y median el proceso de splicing. Este mecanismo fue descubierto en el protozoario ciliado Tetrahymena thermophila. La mayoría de los genes de los vertebrados poseen intrones, a excepción de los genes que codifican para las histonas. Del mismo modo, el número de intrones en un gen puede variar desde unos pocos hasta decenas de estos.8

El splicing puede producir más de un tipo de proteína se llama splicing alternativo—, ya que los exones se arreglan de manera diferencial, creando variedades de ARN mensajeros.8
ARN DE TRANSFERENCIA
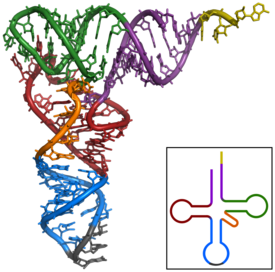
El ARN de transferencia es el encargado de transportar los aminoácidos en el citoplasma para la síntesis de proteínas 9 Esta formado por 70-90 nucleótidos 8La estructura tiene pliegues y cruces (ver figura 30), En uno de los extremos se ubica un anillo adenina, el grupo hidroxilo de la ribosa hace la unión con el aminoácido para ser transportado.8
Los distintos ARN de transferencia se combinan exclusivamente con uno de los veinte aminoácidos que forman las proteínas; El complejo del ARN de transferencia junto con el aminoácido se denomina aminoacil–ARNt.
En el proceso de traducción cada ARN de transferencia reconoce un codón específico en el ARN mensajero. Cuando lo reconoce, el aminoácido correspondiente es liberado y pasa a formar parte del péptido sintetizado, en el emparejamiento de bases entre el ARNt y el ARNm permiten que se inserte el aminoácido correcto en la cadena de polipéptidos presentando fragmentos con estructura de doble hélice y otros en forman bucles, (ver figura 31), el ARN cuenta con un “anticodon” situado en la región media de la molécula. Este anticodón es capaz de formar enlaces de hidrógeno con las bases complementarias presentes en el ADN mensajero.8

MicroARN
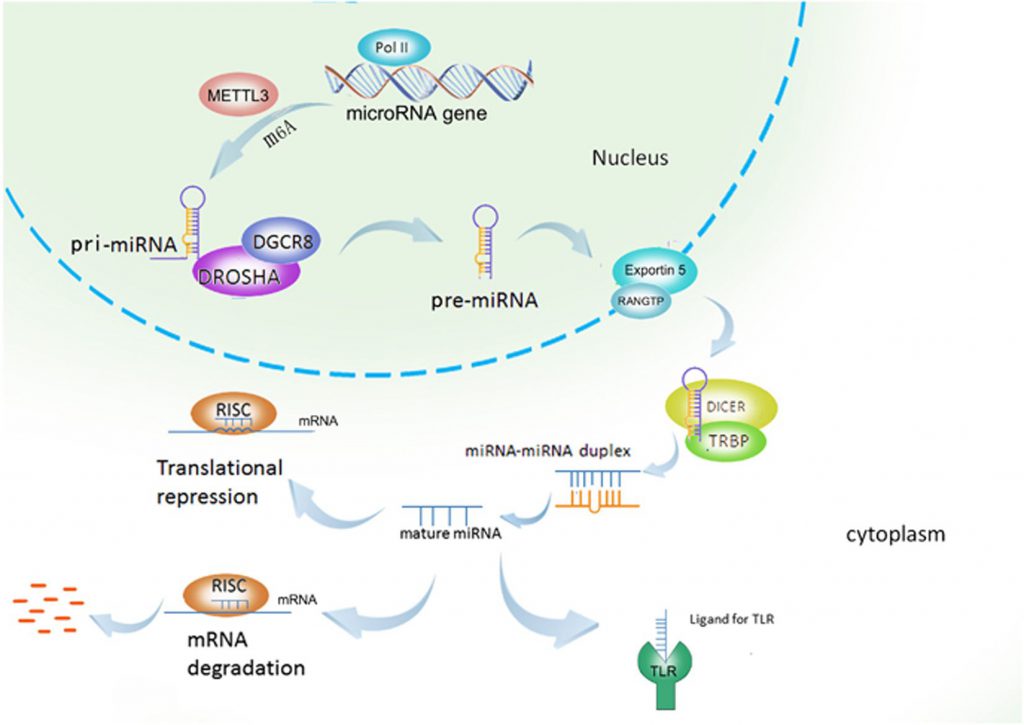
Los micro ARN o ARN mi son de una sola cadena, entre 21 y 23 nucleótidos, cuya función es regular la expresión de los genes.4 Como no se traducen a proteínas, se pueden llamar ARN no codificante.8 Derivan de precursores más largos denominados ARNmi–pri, derivados del primer transcrito del gen. En el núcleo de la célula, estos precursores son modificados en el complejo de microprocesador y el resultado es un pre–ARNmi. Los pre–ARNmi están formados de 70 nucleótidos que continúan su procesamiento en el citoplasma por una enzima llamada Dicer, que ensambla el complejo de silenciamiento inducido por ARN (RISC) y finalmente se sintetiza el ARNmi ( ver figura 32). Estos ARN son capaces de regular la expresión de los genes, ya que son complementarios a ARN mensajeros específicos. 8
ARN DE SILENCIAMIENTO
Es un tipo de micro ARN son cortos de 20 a 25 nucleótidos que obstaculizan la expresión de ciertos genes. A cada codón del ARNm le corresponde el ARNt que posea el anticodón complementario, el cual transporta un determinado aminoácido. Es por eso que cada gen codifica específicamente la síntesis de una proteína, pues proporciona la información necesaria para unir sus aminoácidos en la secuencia adecuada en el proceso de traducción.7
El ARN ribosómico
El ARN ribosómico es el más abundante 80 % del ARN celular. Sus moléculas son largas y monocatenarias, aunque presenta fragmentos con estructura de doble cadena. Tiene función estructural ya que se encuentra con proteínas formando los ribosomas.7
Un ribosoma tiene dos partes: una subunidad grande y una subunidad pequeña, el ARNm se encuentra entre las dos subunidades. Ambas subunidades difieren entre los procariotas y los eucariotas en cuanto al coeficiente de sedimentación. Los procariotas poseen una subunidad grande 50S y una pequeña de 30S, mientras que en los eucariotas la subunidad grande es 60S y la pequeña 40S.8 Los genes que codifican para los ARN ribosomales están en el nucléolo. Los ARN ribosomales son transcritos en una región por la ARN polimerasa I.
Una molécula de ARNt reconoce un codón en el ARNm, se une a él mediante un emparejamiento de bases complementario y agrega el aminoácido correcto a la cadena peptídica en crecimiento.6 El ARNr del ribosoma también tiene una actividad enzimática (peptidil transferasa) y cataliza la formación de los enlaces peptídicos entre dos aminoácidos alineados. La secuencia de bases de ARN es complementaria a la secuencia de codificación del ADN del que se ha copiado. 7
Polirribosomas
Una molécula de ARN mensajero puede dar origen a varias proteínas a la vez, uniéndose a más de un ribosoma. A medida que avanza el proceso de traducción, el extremo del mensajero queda libre y puede ser captado por otro ribosoma, empezando una nueva síntesis. Por eso es común encontrar a los ribosomas agrupados entre 3 y 10, en una sola molécula de ARN mensajero y se le llaman poliribosomas. 8
Referencias
1.- Universidad Nacional de Colombia. (2018). Ácidos Nucleicos. StuDocu.com. Recuperado desde: https://www.studocu.com/es-mx/document/universidad-nacional-de-colombia/colombia-contemporanea/trabajo-de-tutoria/acidos-nucleicos/3966046/view
2.- Burriel, V. (2019). Estructura y Propiedades de los ácidos Nucleicos. Universidad de Valencia. Recuperado desde: https://www.uv.es/tunon/pdf_doc/AcidosNucleicos_veronica.pdf
3.- Rice University. (2017). Biology. OpenStax. Recuperado desde: https://cnx.org/contents/GFy_h8cu@11.10:yxeAKc4X@9/Nucleic-Acids
4.-Centro de Estudios Científicos. (2019). La Historia del ADN. Recursos para la Educación. Recuperado desde: http://www.cecs.cl/educacion/index.php?section=biologia&classe=29&id=58
5. ADN. En: Significados.com. Recuperado desde: https://www.significados.com/adn/Consultado:15 de agosto 2019, 12:01 am.
6.- Khan Academy. (2019). Descubrimiento de la estructura del ADN. Khan Academy. Recuperado desde: https://es.khanacademy.org/science/biology/dna-as-the-genetic-material/dna-discovery-and-structure/a/discovery-of-the-structure-of-dna
7.- Castaños, E. (2019). Estructura y tipos de ARN. Lidia con la Química. Recuperado desde: https://lidiaconlaquimica.wordpress.com/2015/07/20/estructura-y-tipos-de-arn/
8.-Gelambi, M. (2019) ARN: funciones, Estructura y Tipos. Lifeder.com. Recuperado desde: https://www.lifeder.com/arn-rna/
9,. Gelambi, M. (2019) Bases nitrogenadas: clasificación y funciones. Lifeder.com. Recuperado desde: https://www.lifeder.com/bases-nitrogenadas/
10.-EL TIEMPO. (2018). Identifican nueva forma de ADN en células humanas. Eltiempo.com. Recuperado desde: https://www.eltiempo.com/vida/ciencia/i-motif-nueva-forma-de-adn-en-las-celulas-humanas-209332
11. (2013). Descubierta la ´cuádruple hélice´ de ADN en células humanas. Agenciasinc.es. Recuperado desde: https://www.agenciasinc.es/Noticias/Descubierta-la-cuadruple-helice-de-ADN-en-celulas-humanas
12.-Claros, G. (2010). DNA-A o RNA-11. Apuntes de Biología Molecular. Recuperado desde: https://www.sebbm.es/BioROM/contenido/av_bma/apuntes/T3/dnaaz.htm
13.Donoso, J. (2006). Ácidos Nucleicos. Biopolímeros. Recuperado desde: http://facultatciencies.uib.cat/prof/josefa.donoso/campus/modulos/modulo5/modulo5_5_3.htm
14.-admin. (2018). Estructuras de i-motif en el núcleo de las células humanas. AMYS: Asociación de Microbiología y Salud. Recuperado desde: http://www.microbiologiaysalud.org/noticias/estructuras-cuadruples-de-i-motif-en-el-nucleo-d
15.- Berg, J., Tymoczko, J. y Stryer, L. (2002) Biochemistry. W. H. Freeman and Company ISBN 0-7167-4955-6.
16.- Kossel, A.; Steudel, H. Z. (1903). «Weitere Untersuchungen über das Cytosin». Physiol. Chem. 38: 49. doi:10.1515/bchm2.1903.38.1-2.49.
17,. Oró J, Kimball AP (August 1961). “Synthesis of purines under possible primitive earth conditions. I. Adenine from hydrogen cyanide”. Archives of biochemistry and biophysics 94 (2): 217–27. doi:10.1016/0003-9861(61)90033-9. PMID 13731263
18.- wikipedia.org/wiki/Ácido_desoxirribonucleico
19.-Clausen-Schaumann, H., Rief, M., Tolksdorf, C., Gaub, H. (2000). «Mechanical stability of single DNA molecules». Biophys J 78 (4): 1997-2007. PMID 10733978.
20,. Basu, H., Feuerstein, B., Zarling, D., Shafer, R., Marton, L. (1988). «Recognition of Z-RNA and Z-DNA determinants by polyamines in solution: experimental and theoretical studies». J Biomol Struct Dyn 6 (2): 299-309.
21h, D., Kim, Y., Rich, A. (2002). «Z-DNA-binding proteins can act as potent effectors of gene expression in vivo». Proc. Natl. Acad. Sci. U.S.A. 99 (26): 16666-71. PMID 12486233.
22.-Wright, W., Tesmer, V., Huffman, K., Levene, S., Shay, J. (1997). Normal human chromosomes have long G-rich telomeric overhangs at one end. Genes Dev 11 (21): 2801-9. PMID 9353250.
23.- Griffith, J., Comeau, L., Rosenfield, S., Stansel, R., Bianchi, A., Moss, H., De Lange, T. (1999). Mammalian telomeres end in a large duplex loop. Cell 97 (4): 503-14. PMID 10338214.
24.- Watson, J. D.; Baker, T. A.; Bell, S. P.; Gann, A.; Levine, M. y Losick, R (2006). 6. Las estructuras del DNA y el RNA. Biología Molecular del Gen (5ª Ed.) (Madrid: Médica Panamericana). ISBN 84-7903-505-6.
25,.De Robertis, E. D. P. 1998. Biología celular y molecular. El Ateneo, 617 páginas. ISBN 950-02-0364-2. ISBN 978-950-02-0364-7
26,.Valerie, K., Povirk, L. (2003). Regulation and mechanisms of mammalian double-strand break repair. Oncogene 22 (37): 5792-812. PMID 12947387. doi:10.1038/sj.onc.1206679.
27.- Braña, M., Cacho, M., Gradillas, A., de Pascual-Teresa, B., Ramos, A. (2001). Intercalators as anticancer drugs. Curr Pharm Des 7 (17): 1745-80. PMID 11562309. doi:10.2174/1381612013397113.
28.- Bickle, T., Krüger, D. (1993). Biology of DNA restriction. Microbiol Rev 57 (2): 434-50. PMID 8336674.
29.- Doherty, A., Suh, S. (2000). Structural and mechanistic conservation in DNA ligases. Nucleic Acids Res 28 (21): 4051-8. PMID 11058099. doi:10.1093/nar/28.21.4051.
30.- Joyce, C., Steitz, T. (1995) Polymerase structures and function: variations on a theme?. J Bacteriol 177 (22): 6321-9. PMID 7592405.
31.- Nugent C, Lundblad V (1998). The telomerase reverse transcriptase: components and regulation. Genes Dev 12 (8): 1073-85. PMID 9553037.
32.- Avogadro: an open-source molecular builder and visualization tool. Version 1.XX. http://avogadro.cc/